La alineación de secuencias por pares es un proceso en el que se comparan dos secuencias a la vez y se proporciona la mejor alineación de secuencias posible. La alineación de secuencias por pares utiliza un algoritmo de programación dinámica. Biopython tiene un módulo especial Bio.pairwise2 que identifica la secuencia de alineación utilizando el método por pares. Biopython proporciona el mejor algoritmo para encontrar la secuencia de alineación en comparación con otro software.
Tomemos dos secuencias simples e hipotéticas como ejemplo para usar el módulo por pares.
Python3
# Import libraries
from Bio import pairwise2
from Bio.Seq import Seq
# Creating sample sequences
seq1 = Seq("TGTGACTA")
seq2 = Seq("CATGGTCA")
# Finding similarities
alignments = pairwise2.align.globalxx(seq1, seq2)
# Showing results
for match in alignments:
print(match)
Producción:
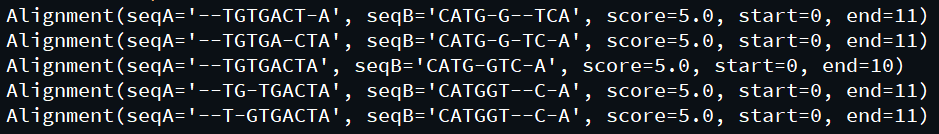
Aquí, el método globalxx hace el trabajo principal, sigue la convención <tipo de alineación>XX donde XX es un código que tiene dos caracteres que indican los parámetros que toma. El primer carácter indica la puntuación de coincidencia y desajuste, mientras que el segundo indica el parámetro para la penalización por brecha.
Parámetros de coincidencia:
| Carácter de código | Descripción |
|---|---|
| X | Sin parámetros. El carácter idéntico tiene una puntuación de 1, de lo contrario 0. |
| metro | puntuación de coincidencia de caracteres idénticos; de lo contrario, puntuación de discrepancia. |
| d | diccionario que devuelve puntuaciones de cualquier par de caracteres. |
| C | Una función de devolución de llamada devuelve puntuaciones. |
Parámetros de penalización por brecha:
| Carácter de código | Descripción |
|---|---|
| X | Sin penalizaciones por hueco. |
| s | ambas secuencias tienen la misma penalización de espacio abierto y extendido. |
| d | Secuencias que tienen diferente penalización de espacio abierto y extendido. |
| C | Una función de devolución de llamada devuelve las penalizaciones de brecha. |
Para una buena impresión , Bio.pairwise2 proporciona el método format_alignment() :
Python3
# Import libraries
from Bio import pairwise2
from Bio.Seq import Seq
from Bio.pairwise2 import format_alignment
# Creating sqmple sequences
seq1 = Seq("TGTGACTA")
seq2 = Seq("CATGGTCA")
# Finding similarities
alignments = pairwise2.align.globalxx(seq1, seq2)
# Showing results
for alignment in alignments:
print(format_alignment(*alignment))
Producción:
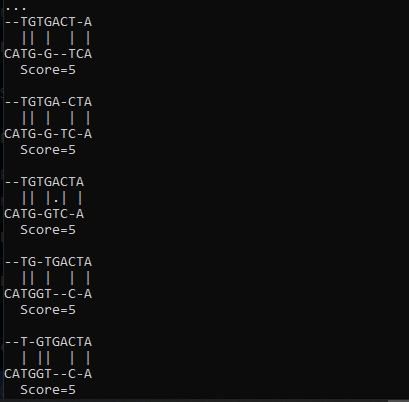
Hay otro módulo proporcionado por Biopython para realizar la alineación de secuencias por pares. El módulo Align tiene un PairwiseAligner() para este propósito. Tiene varias API para establecer parámetros como modo, puntuación de coincidencia, algoritmo, penalización por brecha, etc. A continuación se muestra una implementación simple del método:
Python3
# Import libraries
from Bio import Align
from Bio.Seq import Seq
# Creating sample sequences
seq1 = Seq("TGTGACTA")
seq2 = Seq("CATGGTCA")
# Calling method
aligner = Align.PairwiseAligner()
# Showing method attributes
print(aligner)
# Finding similarities
alignments = aligner.align(seq1, seq2)
# Showing results
for alignment in alignments:
print(alignment)
Producción:
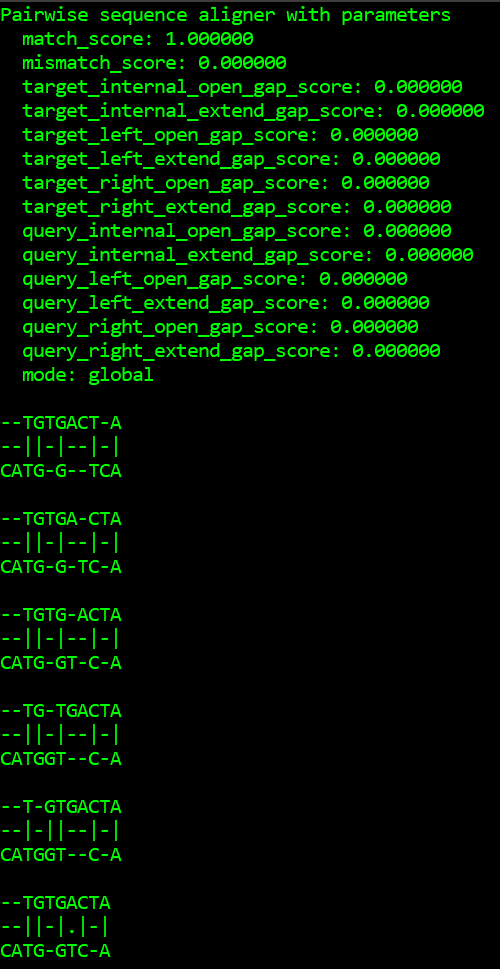
Publicación traducida automáticamente
Artículo escrito por jeeteshgavande30 y traducido por Barcelona Geeks. The original can be accessed here. Licence: CCBY-SA