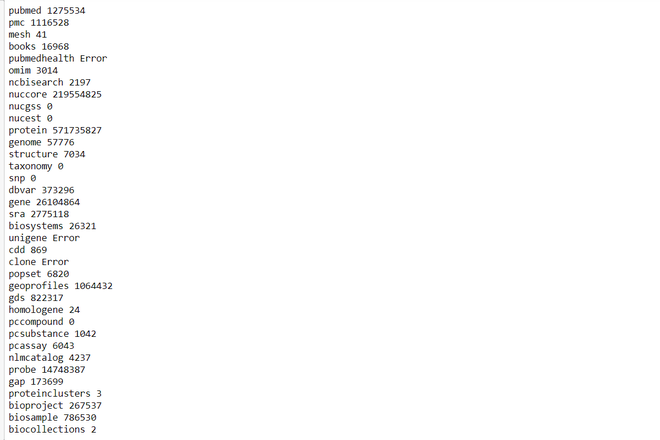El NCBI proporciona un sistema de búsqueda en línea llamado Entrez. Esto proporciona acceso a una amplia gama de bases de datos de biología molecular y también proporciona un sistema de consulta global integrado que admite los operadores booleanos y la búsqueda de campo. Los resultados se devuelven desde todas las bases de datos que contienen información como el número de visitas, enlaces a la base de datos de origen, etc. de cada base de datos.
Funciones utilizadas
Biopython Entrez viene equipado con 2 métodos para realizar operaciones de búsqueda en bases de datos:
- Biopython tiene un método específico de Entrez llamado esearch() para buscar en cualquiera de las bases de datos de Entrez. Acepta la base de datos de parámetros posicionales y el término que tenemos que buscar. Si se asigna una base de datos incorrecta, generará un error.
Sintaxis:
Bio.Entrez. investigación(base de datos, término)
- Para buscar cualquier consulta en todas las bases de datos, se utiliza el método egquery() . Es similar a los métodos de Entrez.esearch() excepto que solo toma el término parámetro omitiendo el parámetro de la base de datos.
Sintaxis:
Bio.Entrez.egquery(término)
Acercarse
- Importe los módulos requeridos.
- Configure su correo electrónico para identificar quién está conectado con la base de datos.
- Configure el parámetro de la herramienta Entrez, es Biopython por defecto.
- Utilice cualquiera de los métodos proporcionados anteriormente con los parámetros apropiados.
- Los datos devueltos estarán en formato XML, por lo que para obtener estos datos en el objeto python, se utiliza el método Entrez.read() para leer el objeto.
- Lea la información proporcionada.
La implementación usando ambos métodos se da a continuación:
Ejemplo 1: Usar essearch()
Python3
# Import libraries from Bio import Entrez # Setting email Entrez.email = 'jeetesh1@yopmail.com' # Setting Entrez tool parameter Entrez.tool = 'Demoscript' # Searching for database info = Entrez.esearch(db="nucleotide", term="genome") # reading records record = Entrez.read(info) # Showing records print(record)
Producción:

Ejemplo 2: Usando egquery()
Python3
# Import libraries from Bio import Entrez # Setting email Entrez.email = 'jeetesh1@yopmail.com' # Setting Entrez tool parameter Entrez.tool = 'Demoscript' # Searching for database info = Entrez.egquery(term="genome") record = Entrez.read(info) for row in record["eGQueryResult"]: print(row["DbName"], row["Count"])
Producción :
Publicación traducida automáticamente
Artículo escrito por jeeteshgavande30 y traducido por Barcelona Geeks. The original can be accessed here. Licence: CCBY-SA