En este artículo veremos cómo podemos resaltar los máximos de imagen en mahotas. Los máximos se pueden encontrar mejor en la imagen del mapa de distancia porque en la imagen etiquetada cada etiqueta es máxima, pero en el mapa de distancia los máximos se pueden identificar fácilmente. Para ello vamos a utilizar la imagen de microscopía fluorescente de un benchmark de segmentación nuclear. Podemos obtener la imagen con la ayuda del comando que se indica a continuación.
mahotas.demos.nuclear_image()
A continuación se muestra la imagen_nuclear
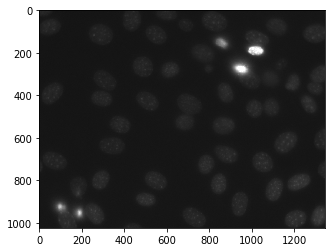
Para hacer esto usaremos el método mahotas.morph.regmax
Sintaxis: mahotas.morph.regmax (img, Bc)
Argumento: toma el objeto de imagen y la array de números como argumento
Retorno: devuelve el objeto de imagen
Nota: la entrada de esto debe ser la imagen filtrada o cargada como gris
. Para filtrar la imagen, tomaremos el objeto de imagen que es numpy.ndarray y lo filtraremos con la ayuda de la indexación, a continuación se muestra el comando para hacer esto.
image = image[:, :, 0]
Ejemplo 1 :
Python3
# importing various libraries
import mahotas
import mahotas.demos
import mahotas as mh
import numpy as np
from pylab import imshow, show
# loading nuclear image
nuclear = mahotas.demos.nuclear_image()
# filtering image
nuclear = nuclear[:, :, 0]
# adding gaussian filter
nuclear = mahotas.gaussian_filter(nuclear, 4)
# setting threshold
threshed = (nuclear > nuclear.mean())
# creating distance map
dmap = mahotas.distance(threshed)
print("Distance Map")
# showing image
imshow(dmap)
show()
# numpy ones array
Bc = np.ones((3, 2))
# getting maxima
maxima = mahotas.morph.regmax(dmap, Bc = Bc)
# showing image
print("Maxima")
imshow(maxima)
show()
Producción :

Ejemplo 2:
Python3
# importing required libraries
import numpy as np
import mahotas
from pylab import imshow, show
# loading image
img = mahotas.imread('dog_image.png')
# filtering the image
img = img[:, :, 0]
# setting gaussian filter
gaussian = mahotas.gaussian_filter(img, 15)
# setting threshold value
gaussian = (gaussian > gaussian.mean())
# creating a labelled image
labelled, n_nucleus = mahotas.label(gaussian)
# getting distance map
dmap = mahotas.distance(labelled)
# showing image
print("Distance Map")
imshow(dmap)
show()
# numpy ones array
Bc = np.ones((4, 1))
# getting maxima
maxima = mahotas.morph.regmax(dmap, Bc = Bc)
# showing image
print("Maxima")
imshow(maxima)
show()
Producción :

Publicación traducida automáticamente
Artículo escrito por rakshitarora y traducido por Barcelona Geeks. The original can be accessed here. Licence: CCBY-SA