El análisis de componentes principales es básicamente un procedimiento estadístico para convertir un conjunto de observaciones de variables posiblemente correlacionadas en un conjunto de valores de variables linealmente no correlacionadas.
Cada uno de los componentes principales se elige de tal manera que describiría la varianza aún disponible de la mayoría de ellos y todos estos componentes principales son ortogonales entre sí. En todos los componentes principales, el primer componente principal tiene una varianza máxima.
Usos de PCA:
- Se utiliza para encontrar la interrelación entre las variables en los datos.
- Se utiliza para interpretar y visualizar datos.
- El número de variables está disminuyendo, lo que simplifica el análisis posterior.
- A menudo se usa para visualizar la distancia genética y la relación entre poblaciones.
Estos se realizan básicamente en una array simétrica cuadrada. Puede ser una array pura de sumas de cuadrados y productos cruzados o una array de covarianza o una array de correlación. Se utiliza una array de correlación si la varianza individual difiere mucho.
Objetivos de PCA:
- Es básicamente un procedimiento no dependiente en el que reduce el espacio de atributos de un gran número de variables a un número menor de factores.
- PCA es básicamente un proceso de reducción de dimensión, pero no hay garantía de que la dimensión sea interpretable.
- La tarea principal en este PCA es seleccionar un subconjunto de variables de un conjunto más grande, en función de qué variables originales tengan la correlación más alta con el monto principal.
Método del eje principal: PCA básicamente busca una combinación lineal de variables para que podamos extraer la máxima varianza de las variables. Una vez que este proceso se completa, lo elimina y busca otra combinación lineal que brinde una explicación sobre la proporción máxima de la varianza restante, lo que básicamente conduce a factores ortogonales. En este método, analizamos la varianza total.
Vector propio: es un vector distinto de cero que permanece paralelo después de la multiplicación de arrays. Supongamos que x es un vector propio de dimensión r de la array M con dimensión r*r si Mx yx son paralelos. Luego necesitamos resolver Mx=Ax donde tanto x como A son desconocidos para obtener el vector propio y los valores propios.
En Eigen-Vectors podemos decir que los componentes principales muestran la varianza común y única de la variable. Básicamente, es un enfoque centrado en la varianza que busca reproducir la varianza total y la correlación con todos los componentes. Los componentes principales son básicamente las combinaciones lineales de las variables originales ponderadas por su contribución para explicar la varianza en una dimensión ortogonal particular.
Valores propios: Se les conoce básicamente como raíces características. Básicamente mide la varianza en todas las variables que se explica por ese factor. La razón de valores propios es la razón de la importancia explicativa de los factores con respecto a las variables. Si el factor es bajo entonces está contribuyendo menos a la explicación de las variables. En palabras simples, mide la cantidad de varianza en la base de datos dada total explicada por el factor. Podemos calcular el valor propio del factor como la suma de su carga factorial al cuadrado para todas las variables.
Ahora, comprendamos el análisis de componentes principales con Python.
Para obtener el conjunto de datos utilizado en la implementación, haga clic aquí .
Paso 1: Importación de las bibliotecas
Python
# importing required libraries import numpy as np import matplotlib.pyplot as plt import pandas as pd
Paso 2: Importación del conjunto de datos
Importe el conjunto de datos y distribúyalo en componentes X e Y para el análisis de datos.
Python
# importing or loading the dataset
dataset = pd.read_csv('wine.csv')
# distributing the dataset into two components X and Y
X = dataset.iloc[:, 0:13].values
y = dataset.iloc[:, 13].values
Paso 3: dividir el conjunto de datos en el conjunto de entrenamiento y el conjunto de prueba
Python
# Splitting the X and Y into the # Training set and Testing set from sklearn.model_selection import train_test_split X_train, X_test, y_train, y_test = train_test_split(X, y, test_size = 0.2, random_state = 0)
Paso 4: Escalado de características
Hacer la parte de preprocesamiento en el conjunto de entrenamiento y prueba, como ajustar la escala estándar.
Python
# performing preprocessing part from sklearn.preprocessing import StandardScaler sc = StandardScaler() X_train = sc.fit_transform(X_train) X_test = sc.transform(X_test)
Paso 5: Aplicación de la función PCA
Aplicación de la función PCA en el conjunto de entrenamiento y prueba para el análisis.
Python
# Applying PCA function on training # and testing set of X component from sklearn.decomposition import PCA pca = PCA(n_components = 2) X_train = pca.fit_transform(X_train) X_test = pca.transform(X_test) explained_variance = pca.explained_variance_ratio_
Paso 6: Ajuste de la regresión logística al conjunto de entrenamiento
Python
# Fitting Logistic Regression To the training set from sklearn.linear_model import LogisticRegression classifier = LogisticRegression(random_state = 0) classifier.fit(X_train, y_train)

Paso 7: Predicción del resultado del conjunto de pruebas
Python
# Predicting the test set result using # predict function under LogisticRegression y_pred = classifier.predict(X_test)
Paso 8: Hacer la array de confusión
Python
# making confusion matrix between # test set of Y and predicted value. from sklearn.metrics import confusion_matrix cm = confusion_matrix(y_test, y_pred)
Paso 9: Predecir el resultado del conjunto de entrenamiento
Python
# Predicting the training set
# result through scatter plot
from matplotlib.colors import ListedColormap
X_set, y_set = X_train, y_train
X1, X2 = np.meshgrid(np.arange(start = X_set[:, 0].min() - 1,
stop = X_set[:, 0].max() + 1, step = 0.01),
np.arange(start = X_set[:, 1].min() - 1,
stop = X_set[:, 1].max() + 1, step = 0.01))
plt.contourf(X1, X2, classifier.predict(np.array([X1.ravel(),
X2.ravel()]).T).reshape(X1.shape), alpha = 0.75,
cmap = ListedColormap(('yellow', 'white', 'aquamarine')))
plt.xlim(X1.min(), X1.max())
plt.ylim(X2.min(), X2.max())
for i, j in enumerate(np.unique(y_set)):
plt.scatter(X_set[y_set == j, 0], X_set[y_set == j, 1],
c = ListedColormap(('red', 'green', 'blue'))(i), label = j)
plt.title('Logistic Regression (Training set)')
plt.xlabel('PC1') # for Xlabel
plt.ylabel('PC2') # for Ylabel
plt.legend() # to show legend
# show scatter plot
plt.show()
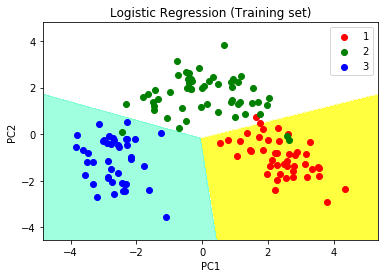
Paso 10: visualización de los resultados del conjunto de pruebas
Python
# Visualising the Test set results through scatter plot
from matplotlib.colors import ListedColormap
X_set, y_set = X_test, y_test
X1, X2 = np.meshgrid(np.arange(start = X_set[:, 0].min() - 1,
stop = X_set[:, 0].max() + 1, step = 0.01),
np.arange(start = X_set[:, 1].min() - 1,
stop = X_set[:, 1].max() + 1, step = 0.01))
plt.contourf(X1, X2, classifier.predict(np.array([X1.ravel(),
X2.ravel()]).T).reshape(X1.shape), alpha = 0.75,
cmap = ListedColormap(('yellow', 'white', 'aquamarine')))
plt.xlim(X1.min(), X1.max())
plt.ylim(X2.min(), X2.max())
for i, j in enumerate(np.unique(y_set)):
plt.scatter(X_set[y_set == j, 0], X_set[y_set == j, 1],
c = ListedColormap(('red', 'green', 'blue'))(i), label = j)
# title for scatter plot
plt.title('Logistic Regression (Test set)')
plt.xlabel('PC1') # for Xlabel
plt.ylabel('PC2') # for Ylabel
plt.legend()
# show scatter plot
plt.show()
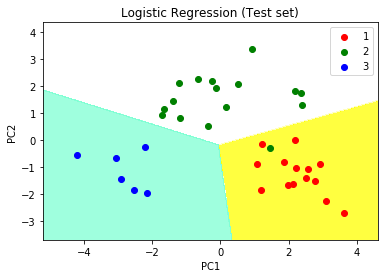
Publicación traducida automáticamente
Artículo escrito por Akashkumar17 y traducido por Barcelona Geeks. The original can be accessed here. Licence: CCBY-SA