Requisitos previos: agrupación en clústeres de K-Means
Un paso fundamental para cualquier algoritmo no supervisado es determinar el número óptimo de clústeres en los que se pueden agrupar los datos. El método del codo es uno de los métodos más populares para determinar este valor óptimo de k.
Ahora demostramos el método dado usando la técnica de agrupamiento de K-Means usando la biblioteca Sklearn de python.
Paso 1: Importación de las bibliotecas requeridas
Python3
from sklearn.cluster import KMeans from sklearn import metrics from scipy.spatial.distance import cdist import numpy as np import matplotlib.pyplot as plt
Paso 2: Crear y visualizar los datos
Python3
# Creating the data
x1 = np.array([3, 1, 1, 2, 1, 6, 6, 6, 5, 6, 7, 8, 9, 8, 9, 9, 8])
x2 = np.array([5, 4, 5, 6, 5, 8, 6, 7, 6, 7, 1, 2, 1, 2, 3, 2, 3])
X = np.array(list(zip(x1, x2))).reshape(len(x1), 2)
# Visualizing the data
plt.plot()
plt.xlim([0, 10])
plt.ylim([0, 10])
plt.title('Dataset')
plt.scatter(x1, x2)
plt.show()
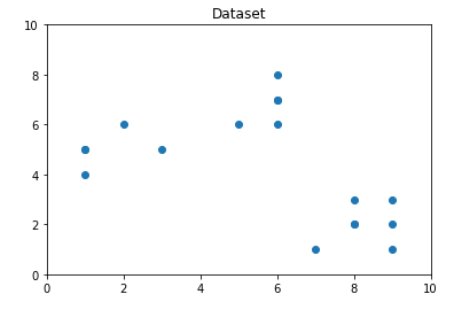
A partir de la visualización anterior, podemos ver que la cantidad óptima de clústeres debe ser alrededor de 3. Pero la visualización de los datos por sí sola no siempre puede dar la respuesta correcta. Por lo tanto, demostramos los siguientes pasos.
Ahora definimos lo siguiente:
- Distorsión: se calcula como el promedio de las distancias al cuadrado desde los centros de los clústeres respectivos. Por lo general, se utiliza la métrica de distancia euclidiana.
- Inercia: Es la suma de las distancias al cuadrado de las muestras a su centro de conglomerado más cercano.
Iteramos los valores de k de 1 a 9 y calculamos los valores de distorsiones para cada valor de k y calculamos la distorsión y la inercia para cada valor de k en el rango dado.
Paso 3: Construcción del modelo de agrupamiento y cálculo de los valores de Distorsión e Inercia:
Python3
distortions = []
inertias = []
mapping1 = {}
mapping2 = {}
K = range(1, 10)
for k in K:
# Building and fitting the model
kmeanModel = KMeans(n_clusters=k).fit(X)
kmeanModel.fit(X)
distortions.append(sum(np.min(cdist(X, kmeanModel.cluster_centers_,
'euclidean'), axis=1)) / X.shape[0])
inertias.append(kmeanModel.inertia_)
mapping1[k] = sum(np.min(cdist(X, kmeanModel.cluster_centers_,
'euclidean'), axis=1)) / X.shape[0]
mapping2[k] = kmeanModel.inertia_
Paso 4: Tabulación y Visualización de los resultados
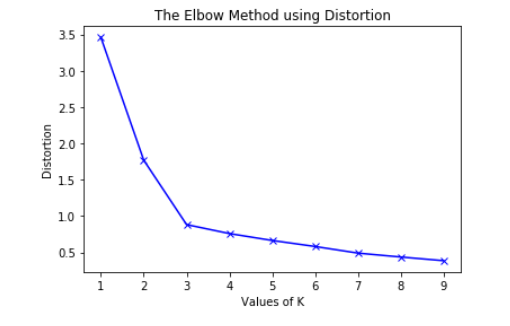
a) Utilizando los diferentes valores de Distorsión:
Python3
for key, val in mapping1.items():
print(f'{key} : {val}')

Python3
plt.plot(K, distortions, 'bx-')
plt.xlabel('Values of K')
plt.ylabel('Distortion')
plt.title('The Elbow Method using Distortion')
plt.show()
b) Usando los diferentes valores de Inercia:
Python3
for key, val in mapping2.items():
print(f'{key} : {val}')

Python3
plt.plot(K, inertias, 'bx-')
plt.xlabel('Values of K')
plt.ylabel('Inertia')
plt.title('The Elbow Method using Inertia')
plt.show()
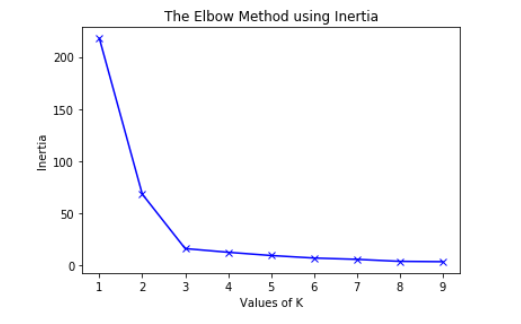
Para determinar el número óptimo de grupos, tenemos que seleccionar el valor de k en el «codo», es decir, el punto después del cual la distorsión/inercia comienza a disminuir de forma lineal. Por lo tanto, para los datos dados, concluimos que el número óptimo de conglomerados para los datos es 3 .
Los puntos de datos agrupados para diferentes valores de k:-
1. k = 1
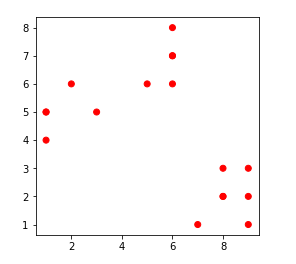
2. k = 2
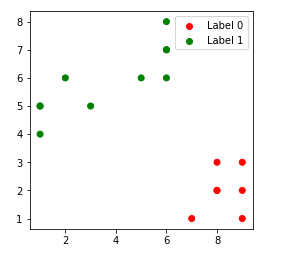
3. k = 3
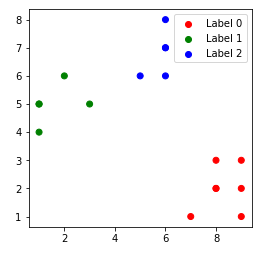
4. k = 4
Publicación traducida automáticamente
Artículo escrito por AlindGupta y traducido por Barcelona Geeks. The original can be accessed here. Licence: CCBY-SA
